毕业设计:基于机器学习的寄生虫微生物图象识别与分类系统 计算机视觉 深度学习 YOLO
毕业设计:基于深度学习的医学图像中寄生虫卵自动识别与分类系统收集并构建了包含多种寄生虫卵的医学图像数据集,并进行了图像预处理和数据增强,以提高模型的训练效果。随后,采用卷积神经网络(CNN)作为基础模型,并结合迁移学习,利用预训练模型对寄生虫卵进行特征提取和分类。通过对模型参数的优化和训练,系统能够在不同种类的寄生虫卵识别任务中实现高效和高准确率。
目录
前言
📅大四是整个大学期间最忙碌的时光,一边要忙着备考或实习为毕业后面临的就业升学做准备,一边要为毕业设计耗费大量精力。近几年各个学校要求的毕设项目越来越难,有不少课题是研究生级别难度的,对本科同学来说是充满挑战。为帮助大家顺利通过和节省时间与精力投入到更重要的就业和考试中去,学长分享优质的选题经验和毕设项目与技术思路。
🚀对毕设有任何疑问都可以问学长哦!
大家好,这里是海浪学长计算机毕设专题,本次分享的课题是
🎯基于机器学习的寄生虫微生物图象识别与分类系统
项目背景
寄生虫感染是全球范围内严重的公共卫生问题,尤其在发展中国家,对人们的健康构成了重大威胁。寄生虫卵的早期识别与分类对于疾病的预防和治疗至关重要。传统的寄生虫卵识别方法依赖于显微镜下的人工观察,效率低且容易受到人为因素的影响。随着深度学习技术的快速发展,利用计算机视觉和深度学习模型自动识别和分类医学图像中的寄生虫卵,成为提升检测效率和准确性的有效手段。因此,开发一套基于深度学习的寄生虫卵自动识别与分类系统,具有重要的临床应用价值和社会意义。
数据集
数据采集时,采用了两种主要的图像采集方法。首先,通过监控视频和相机拍摄特定的护目镜样本,虽然可以获取一些样本,但由于数据集中相同类型图像的比例较高,导致广泛性和代表性不足,从而增加了模型过拟合的风险。其次,通过互联网搜索引擎指定关键字搜索相关护目镜图像并利用爬虫工具自动下载,这一方法确保了图像数量的充足和多样性,显著提高了数据集制作效率。
使用Labelme工具进行图像标注是一种高效且实用的方法。Labelme允许用户对图像中的感兴趣区域进行精确标注,能够快速生成相应的标注文件,这对于后续模型训练至关重要。标注完成后,数据集需要进行合理的划分,通常分为训练集、验证集和测试集,以确保模型在不同数据上的学习和评估效果。通常比例为70%训练集、15%验证集和15%测试集。
from sklearn.model_selection import train_test_split
# 示例代码:划分数据集
X_train, X_temp, y_train, y_temp = train_test_split(X, y, test_size=0.3, random_state=42)
X_val, X_test, y_val, y_test = train_test_split(X_temp, y_temp, test_size=0.5, random_state=42)为了增强数据集的多样性和鲁棒性,数据扩展技术也不可或缺。通过对原始图像进行旋转、平移、缩放、翻转等操作,可以生成更多样本,从而增加模型的泛化能力。这些扩展操作能够帮助模型更好地适应不同的输入情况,提高其在实际应用中的表现。通过结合标注、划分和数据扩展的过程,可以构建出一个高质量的微生物图象数据集
设计思路
数学形态学是一种处理图像的工具,主要依赖于形状和结构的分析。通过应用形态学操作,如膨胀、腐蚀、打开和关闭等,可以高效地分离图像中的寄生虫特征与背景,去除杂质和噪声。这种方法能够提取出寄生虫的边缘和结构特征,为后续的分类任务打下坚实的基础。通过这种图像预处理,系统能够显著提高对寄生虫的识别精度。

在特征提取和选择的过程中,系统会提取出寄生虫图像中的重要特征,这些特征可能包括形态特征、纹理特征和颜色特征等。特征筛选步骤旨在减少冗余信息,提高分类模型的效率和准确性。通过运用一些特征选择算法,如主成分分析(PCA)或线性判别分析(LDA),系统能够识别出对分类任务最具判别能力的特征。这种特征选择的过程能够有效降低数据维度,减少计算复杂性,同时增强模型的泛化能力。

采用改进后的K近邻分类器(KNN)进行寄生虫的识别与分类。KNN是一种简单而有效的非参数分类方法,通过计算待分类样本与训练样本之间的距离,将待分类样本分配给其最近的K个邻居的类别。改进后的KNN算法可以结合加权机制或集成学习的思想,使得分类结果更加准确。此外,KNN对数据分布的敏感性和局部特征的捕捉能力,使其在处理高维特征时表现出色。

在微生物图像识别与分类系统中,采用改进的 K 近邻分类器(KNN)对提取的特征参数进行识别和分类是至关重要的。为了确保分类的准确性,首先需对所有特征参数进行归一化处理,使其取值范围在 0 到 1 之间。这一过程可以消除各特征参数之间的量纲差异,确保在计算样本之间的距离时,各特征的影响力是均匀的。此外,考虑到不同特征在分类过程中的贡献度不同,采用特征加权算法来赋予各特征参数不同的权值,以便更好地反映其对分类结果的影响。
特征权值的计算步骤如下:
- 采用传统的 KNN 分类方法对多个测试样本进行测试,并统计与实际分类之间的错误个数。
- 逐一去除样本中的每个特征参数,使用 KNN 分类,并记录去除特征后的错误分类样本数量。
- 根据错误分类样本数的比值,评估去除特征对分类结果的影响,确定每个特征参数的贡献度。
- 利用这些贡献度计算出各特征的权值,以便在后续的距离计算中加权考虑。
# 示例代码:特征权值计算
import numpy as np
m = original_classification_errors # 原始错误分类个数
m_k = [] # 存储去除特征后错误分类个数
for k in range(1, N+1):
# 去除第 k 个特征后进行分类并统计错误
m_k.append(classification_with_removed_feature(k))
p_k = [mk / m for mk in m_k] # 计算 p_k
weights = [1 / (1 + pk) for pk in p_k] # 计算特征权值使用 K-均值聚类算法来处理数据集,以确保选择到的训练样本具有代表性并且差异性较大。首先,从图像中选取同一类型的微生物样本,设定一个阈值,当某个样本的特征向量与所有聚类中心的最小距离值大于该阈值时,该样本将作为新的聚类中心。通过这种方式,能够有效去除相似样本,降低数据集的冗余。
# 示例代码:K-均值聚类算法
from sklearn.cluster import KMeans
# 假设 features 是微生物样本的特征向量
kmeans = KMeans(n_clusters=num_classes, random_state=42).fit(features)
cluster_centers = kmeans.cluster_centers_ # 获取聚类中心在完成特征加权和数据集的准备后,可以利用改进后的 KNN 算法进行微生物的识别与分类。计算测试样本的特征向量与数据集中每个样本的特征向量之间的加权距离。对计算出的距离进行排序,取出最小距离的样本。如果该距离超过拒判阈值,则判定该对象不属于任何已知微生物类别;否则,进入下一步。取出 K 个最小距离对应的类别编号,并计算同种类别编号中的距离倒数之和,距离倒数之和最小的类别即为待测试对象的分类结果。
海浪学长项目示例:
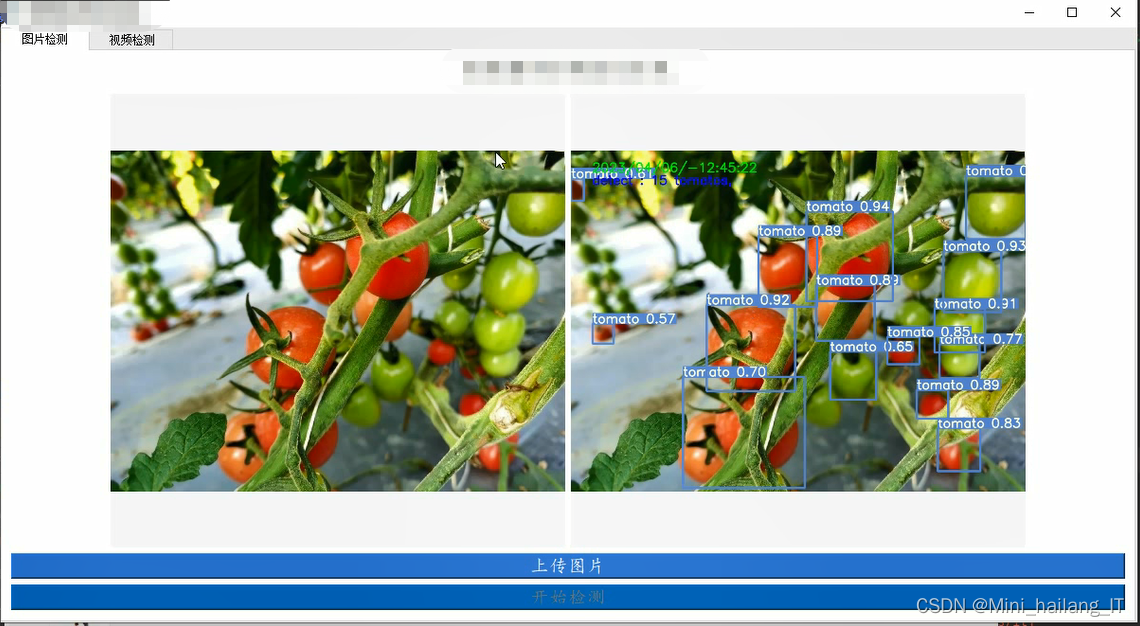



更多帮助
更多推荐
 已为社区贡献65条内容
已为社区贡献65条内容










所有评论(0)