MachineLearning 22. 机器学习之有监督主成分分析筛选基因 (SuperPC)
简介有监督的主成分回归的基本原理主成分分析是由KarlPearson于1901提出的,它把原来的多个变量用少数几个线性组合,即主成分来概括。主成分分析也是降维方法的一种,可以用于可能高度相关的基因表达数据的探索,但是由于在仅考虑自变量的常规主成分分析中组成和选择主成分的时候没有用到生存时间,因此无法保证所选择的主成分与病人的生存相关。所以Bair和Tibshirani提出了有监督的主...

简 介
有监督的主成分回归的基本原理
主成分分析是由KarlPearson于1901提出的,它把原来的多个变量用少数几个线性组合,即主成分来概括。主成分分析也是降维方法的一种,可以用于可能高度相关的基因表达数据的探索,但是由于在仅考虑自变量的常规主成分分析中组成和选择主成分的时候没有用到生存时间,因此无法保证所选择的主成分与病人的生存相关。所以Bair和Tibshirani提出了有监督的主成分分析方法。SuperPC是对常规主成分分析的一种改进。这种方法在降维的时候考虑了生存时间,其核心思想就是只对与生存时间密切相关的基因进行主成分分析。
有监督的主成分过程可以看成是一个识别有关预报因子的聚类的最简单的方法:
a) 根据单变量筛选来选择与生存密切相关的基因,以去除变异的无关来源;
b)应用主成分来识别共同表达基因的分组。

分析步骤
假设有N个观察单位,测量了p个基因,令X为nxp的基因表达矩阵,y为n维的结果向量。假设结果是一个包含截尾数据的生存时间。令x表示第j个病人的第i个基因的表达水平。对此类数据进行生存预测,有监督主成分分析的分析步骤概括如下:
1.计算每一个基因与生存的关系。
2.根据单基因的似然比检验挑选基因,组成简化矩阵。
3.对简化矩阵进行主成分分析,提取一个(或前几个)主成分。
4.将提取出来的主成分作为Cox比例风险模型的协变量拟合模型。
5.根据拟合好的模型对新得到的基因数据集进行生存预测。
软件包安装
superpc 软件包还是存在一些依赖包,正常安装就可以了,主要一下 BiocManager 版本为 3.18,升级一下就好了。
chooseBioCmirror()
install.packages('tidyverse')
BiocManager::install (version ="3.18")
BiocManager::install("survcomp",version ='3.18')
BiocManager::version()
install.packages("superpc")
install.packages("devtools")
library("devtools")
devtools::install_github("jedazard/superpc")
install.packages('survival')
install.packages("glmnet")
install.packages("sampling")数据读取
我们还是利用TCGA-LUAD 的数据集,然后对数据进行处理,分割数据为测试集和验证集,如下:
library(superpc)
library(survival)
library(survcomp)
library(tidyverse)
library(glmnet)
clin <- read.table("clinical.txt", header = T)
head(clin)
## Tumor_Sample_Barcode gender vital_status status time
## 1 TCGA-56-7580 MALE Alive 1 40
## 2 TCGA-58-A46N MALE Alive 1 540
## 3 TCGA-56-7730 MALE Alive 1 6
## 4 TCGA-58-A46M MALE Alive 1 707
## 5 TCGA-85-A4JC MALE Alive 1 -10
## 6 TCGA-37-3783 MALE Alive 1 122
rpkm <- as.matrix(read.table("TCGA_LUSC.m6A_gene.exp.txt", header = T, row.names = 1))
head(rpkm[1:5, 1:5])
## TCGA.77.A5GA.01A TCGA.58.8387.01A TCGA.22.4599.01A TCGA.77.7142.11A
## YTHDC2 10.241983 9.497852 8.951285 10.53625
## IGF2BP2 8.285402 11.725792 11.398209 10.03617
## YTHDC1 10.854868 11.413628 9.914385 12.18859
## ALKBH5 12.409921 11.083479 10.727070 12.31515
## HNRNPC 13.510764 14.808411 13.868533 13.42062
## TCGA.NC.A5HJ.01A
## YTHDC2 10.57365
## IGF2BP2 11.58449
## YTHDC1 11.95529
## ALKBH5 12.32024
## HNRNPC 14.14131
colnames(rpkm) = gsub("[.]", "-", substring(colnames(rpkm), 1, 12))
rpkm = unique(rpkm)
rpkm = as.data.frame(t(rpkm))
rownames(rpkm) = gsub("[.]", "-", rownames(rpkm))
rpkm$Tumor_Sample_Barcode = rownames(rpkm)
lung = merge(clin, rpkm, by.x = "Tumor_Sample_Barcode", by.y = "Tumor_Sample_Barcode")
head(lung)
## Tumor_Sample_Barcode gender vital_status status time YTHDC2 IGF2BP2
## 1 TCGA-18-3406 MALE Dead 2 371 10.490851 8.870365
## 2 TCGA-18-3407 MALE Dead 2 136 11.032735 8.603626
## 3 TCGA-18-3408 FEMALE Alive 1 2099 9.796040 11.279611
## 4 TCGA-18-3409 MALE Alive 1 2417 9.856426 12.023061
## 5 TCGA-18-3410 MALE Dead 2 146 11.074810 6.584963
## 6 TCGA-18-3411 FEMALE Alive 1 1415 9.881114 12.384514
## YTHDC1 ALKBH5 HNRNPC FMR1 HNRNPA2B1 ZC3H13 IGF2BP3 FTO
## 1 10.69870 10.68738 14.38485 10.59059 13.69675 10.23122 6.523562 10.58684
## 2 12.28511 11.84823 14.44876 11.49935 15.05431 11.94361 9.579316 11.26268
## 3 11.57128 12.10165 14.67325 11.72877 14.47168 11.24911 10.005625 11.33204
## 4 11.92258 12.32839 14.16034 11.19168 14.44320 11.83368 4.954196 11.58825
## 5 11.98762 12.39714 14.92435 11.17555 15.51209 11.36632 9.786270 10.91289
## 6 11.56701 12.23751 15.74026 10.48784 15.16632 10.94910 10.558421 11.18177
## METTL14 WTAP YTHDF1 IGF2BP1 RBM15 KIAA1429 METTL3 YTHDF3
## 1 9.063395 13.19583 10.61102 2.584963 8.164907 10.85720 8.924813 12.03032
## 2 9.552669 12.67331 12.07046 1.584963 9.087463 11.58543 10.689124 12.26004
## 3 9.949827 12.61218 11.92518 2.584963 8.280771 11.45224 10.134426 12.23182
## 4 10.675957 12.28135 11.25798 2.321928 9.038919 12.19476 10.029287 12.48734
## 5 10.990104 14.66217 11.67375 7.643856 9.594325 12.09902 10.813781 12.80272
## 6 9.914385 12.97549 11.51668 11.499348 9.204571 12.02721 11.310613 12.48382
## YTHDF2
## 1 11.31175
## 2 12.31175
## 3 11.53916
## 4 12.17773
## 5 12.20427
## 6 11.96939数据分割
library(sampling)table(lung$status)##
## 1 2
## 343 158lung$status <- ifelse(lung$status == 2, 1, 0)
lung <- na.omit(lung) # 去掉NA
set.seed(123)
# 每层抽取70%的数据
train_id <- strata(lung, "status", size = rev(round(table(lung$status) * 0.7)))$ID_unit# 训练数据
trainData <- lung[train_id, ]
# 测试数据
testData <- lung[-train_id, ]构造数据对象,以便读取变量:
# 准备预测变量和结果变量
train.x <- as.matrix(t(trainData[, 6:24]))
# 设置y,把删失的改成负值
train.y <- trainData$time
featurenames = colnames(trainData[, 6:24])
featurenames
## [1] "YTHDC2" "IGF2BP2" "YTHDC1" "ALKBH5" "HNRNPC" "FMR1"
## [7] "HNRNPA2B1" "ZC3H13" "IGF2BP3" "FTO" "METTL14" "WTAP"
## [13] "YTHDF1" "IGF2BP1" "RBM15" "KIAA1429" "METTL3" "YTHDF3"
## [19] "YTHDF2"
data1 = list(x = train.x, y = train.y, censoring.status = trainData$status, featurenames = featurenames)实例操作
构建有监督主成分模型
fit <- superpc.train(data = data1, type = "survival")交叉验证
cv.fit <- superpc.cv(fit, data1, n.threshold = 20, #default
n.fold = 10,
n.components = 3,
min.features = 5,
max.features = nrow(data1$x),
compute.fullcv = TRUE,
compute.preval = TRUE)
superpc.plotcv(cv.fit)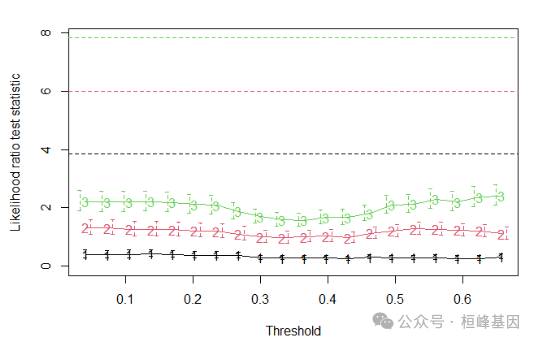
验证集验证
test.x <- as.matrix(t(testData[, 6:24]))
# 设置y,把删失的改成负值
test.y <- testData$time
featurenames = colnames(testData[, 6:24])
featurenames
## [1] "YTHDC2" "IGF2BP2" "YTHDC1" "ALKBH5" "HNRNPC" "FMR1"
## [7] "HNRNPA2B1" "ZC3H13" "IGF2BP3" "FTO" "METTL14" "WTAP"
## [13] "YTHDF1" "IGF2BP1" "RBM15" "KIAA1429" "METTL3" "YTHDF3"
## [19] "YTHDF2"
data2 = list(x = test.x, y = test.y, censoring.status = testData$status, featurenames = featurenames)
superpc.predictionplot(fit, data1, data2, threshold = 1, n.components = 2)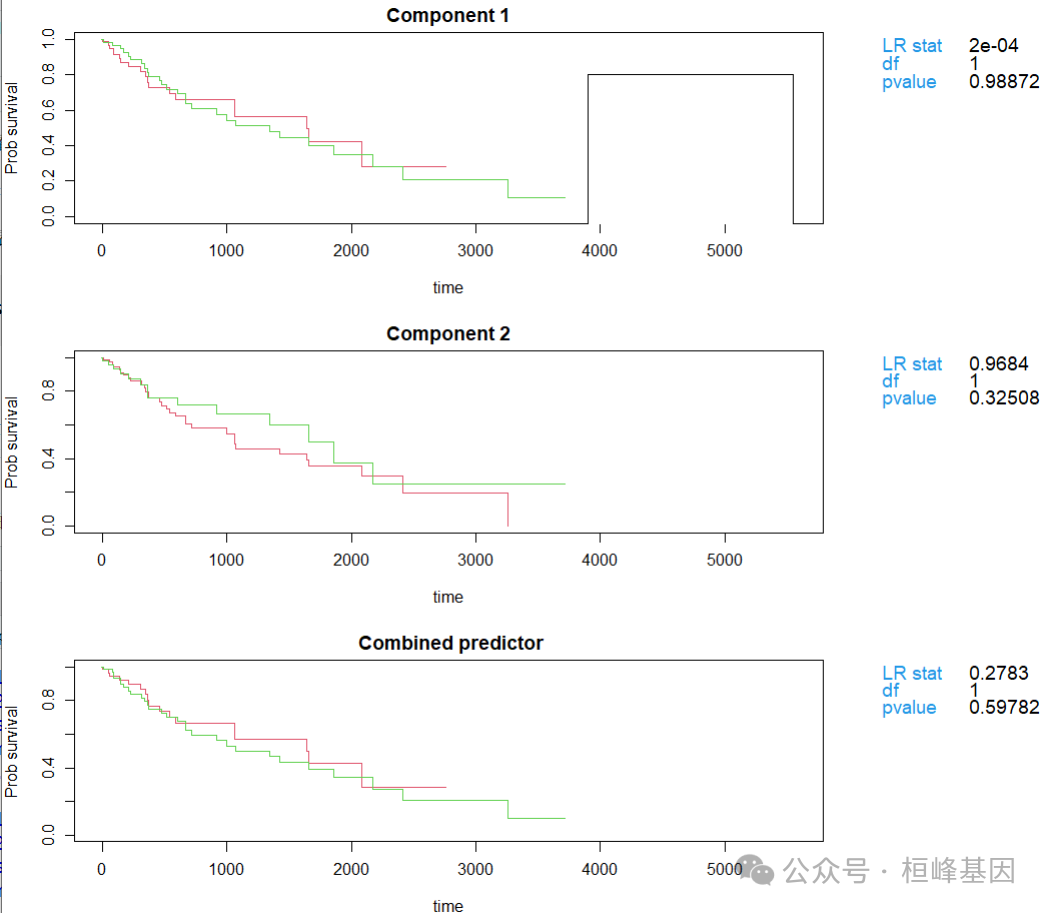
绘制彩虹预测结果
pred <- superpc.predict(fit, data1, data2, threshold = 0.25, n.components = 1)$v.pred
competing.predictors.test <- list(pred1 = rnorm(147), pred2 = as.factor(sample(c(1,
2), replace = TRUE, size = 147)))
sample.labels <- paste("te", as.character(1:147), sep = "")
superpc.rainbowplot(data1, pred, sample.labels, competing.predictors = competing.predictors.test)
## NULL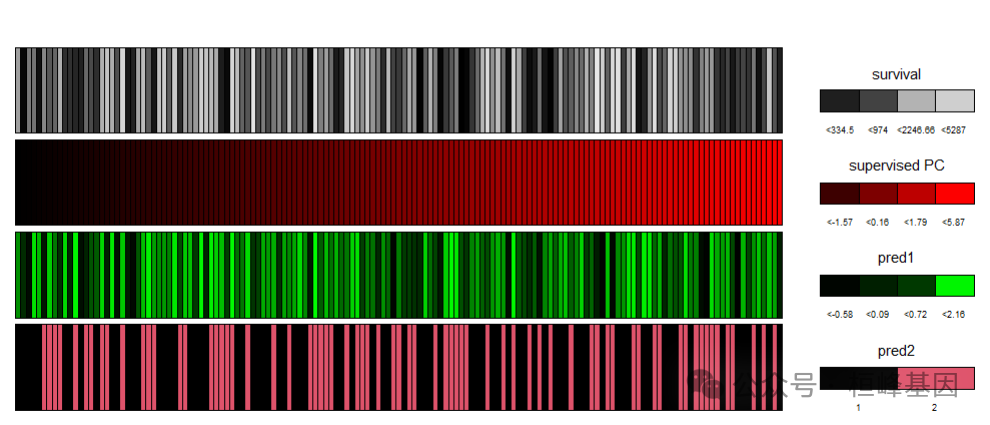
计算一致性 C-index
testData$pred = pred
C_index <- data.frame(Cindex = as.numeric(summary(coxph(Surv(time, status) ~ pred,
testData))$concordance[1]))
C_index
## Cindex
## 1 0.5224172Reference
-
Bair E. and Tibshirani R. Semi-supervised methods to predict patient survival from gene expression data. PLoS Biol (2004), 2(4):e108.
-
Bair E., Hastie T., Paul D., and Tibshirani R. Prediction by supervised principal components. J. Am. Stat. Assoc (2006), 101(473):119-137.
基于机器学习构建临床预测模型
MachineLearning 2. 因子分析(Factor Analysis)
MachineLearning 3. 聚类分析(Cluster Analysis)
MachineLearning 4. 癌症诊断方法之 K-邻近算法(KNN)
MachineLearning 5. 癌症诊断和分子分型方法之支持向量机(SVM)
MachineLearning 6. 癌症诊断机器学习之分类树(Classification Trees)
MachineLearning 7. 癌症诊断机器学习之回归树(Regression Trees)
MachineLearning 8. 癌症诊断机器学习之随机森林(Random Forest)
MachineLearning 9. 癌症诊断机器学习之梯度提升算法(Gradient Boosting)
MachineLearning 10. 癌症诊断机器学习之神经网络(Neural network)
MachineLearning 11. 机器学习之随机森林生存分析(randomForestSRC)
MachineLearning 12. 机器学习之降维方法t-SNE及可视化(Rtsne)
MachineLearning 13. 机器学习之降维方法UMAP及可视化 (umap)
MachineLearning 14. 机器学习之集成分类器(AdaBoost)
MachineLearning 15. 机器学习之集成分类器(LogitBoost)
MachineLearning 16. 机器学习之梯度提升机(GBM)
MachineLearning 17. 机器学习之围绕中心点划分算法(PAM)
MachineLearning 18. 机器学习之贝叶斯分析类器(Naive Bayes)
MachineLearning 19. 机器学习之神经网络分类器(NNET)
MachineLearning 20. 机器学习之袋装分类回归树(Bagged CART)
号外号外,桓峰基因单细胞生信分析免费培训课程即将开始快来报名吧!
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
桓峰基因官网正式上线,请大家多多关注,还有很多不足之处,大家多多指正!http://www.kyohogene.com/
桓峰基因和投必得合作,文章润色优惠85折,需要文章润色的老师可以直接到网站输入领取桓峰基因专属优惠券码:KYOHOGENE,然后上传,付款时选择桓峰基因优惠券即可享受85折优惠哦!https://www.topeditsci.com/

更多推荐
 已为社区贡献25条内容
已为社区贡献25条内容









所有评论(0)