Python实现SMA黏菌优化算法优化循环神经网络分类模型(LSTM分类算法)项目实战
Python实现SMA黏菌优化算法优化循环神经网络分类模型(LSTM分类算法)项目实战
说明:这是一个机器学习实战项目(附带数据+代码+文档+视频讲解),如需数据+代码+文档+视频讲解可以直接到文章最后获取。


1.项目背景
黏菌优化算法(Slime mould algorithm,SMA)由Li等于2020年提出,其灵感来自于黏菌的扩散和觅食行为,属于元启发算法。具有收敛速度快,寻优能力强的特点。主要模拟了黏菌的扩散及觅食行为,利用自适应权重模拟了基于生物振荡器的“黏菌传播波”产生正反馈和负反馈的过程,形成具有良好的探索能力和开发倾向的食物最优连接路径,因此具有较好的应用前景。
本项目通过SMA黏菌优化算法优化循环神经网络分类模型。
2.数据获取
本次建模数据来源于网络(本项目撰写人整理而成),数据项统计如下:
|
编号 |
变量名称 |
描述 |
|
1 |
x1 |
|
|
2 |
x2 |
|
|
3 |
x3 |
|
|
4 |
x4 |
|
|
5 |
x5 |
|
|
6 |
x6 |
|
|
7 |
x7 |
|
|
8 |
x8 |
|
|
9 |
x9 |
|
|
10 |
x10 |
|
|
11 |
y |
因变量 |
数据详情如下(部分展示):
3.数据预处理
3.1 用Pandas工具查看数据
使用Pandas工具的head()方法查看前五行数据:
关键代码:

3.2数据缺失查看
使用Pandas工具的info()方法查看数据信息:
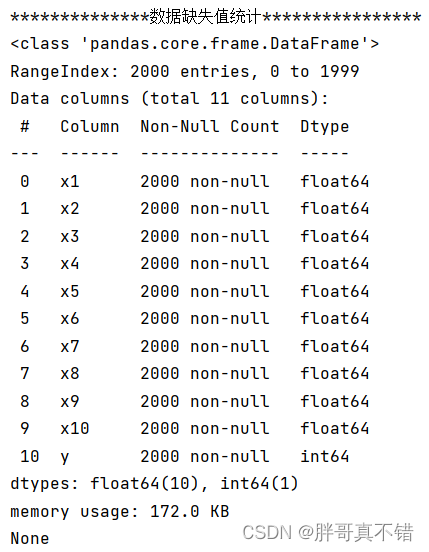
从上图可以看到,总共有11个变量,数据中无缺失值,共2000条数据。
关键代码:

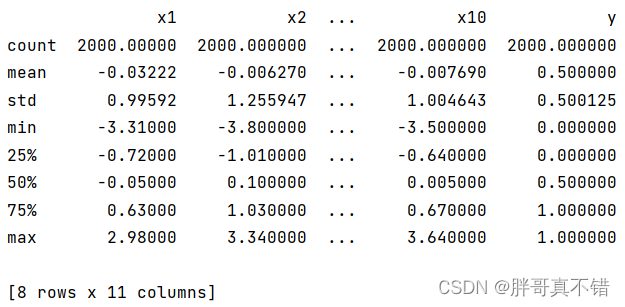
3.3数据描述性统计
通过Pandas工具的describe()方法来查看数据的平均值、标准差、最小值、分位数、最大值。
关键代码如下:

4.探索性数据分析
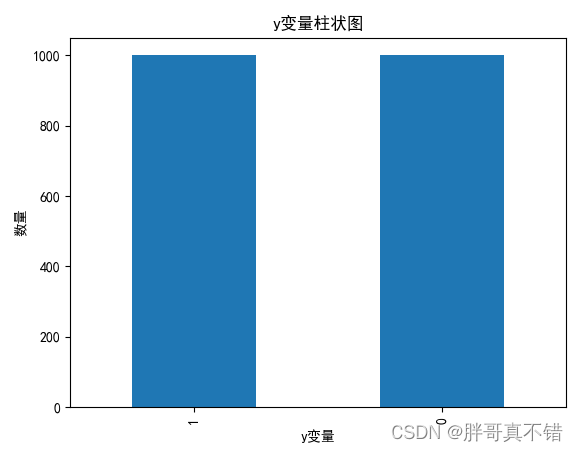
4.1 y变量柱状图
用Matplotlib工具的plot()方法绘制柱状图:
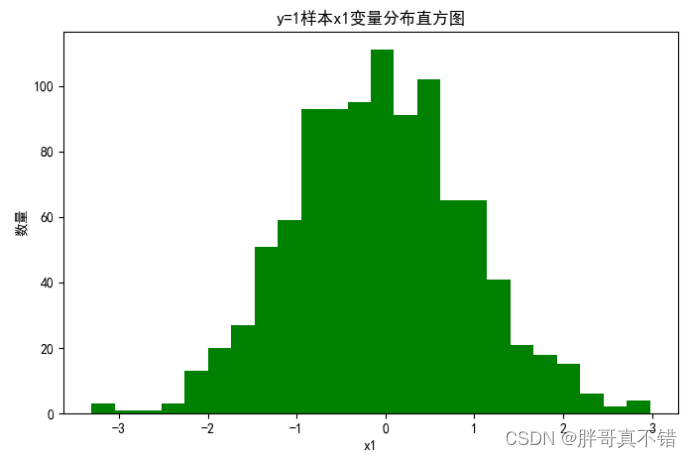
4.2 y=1样本x1变量分布直方图
用Matplotlib工具的hist()方法绘制直方图:
4.3 相关性分析
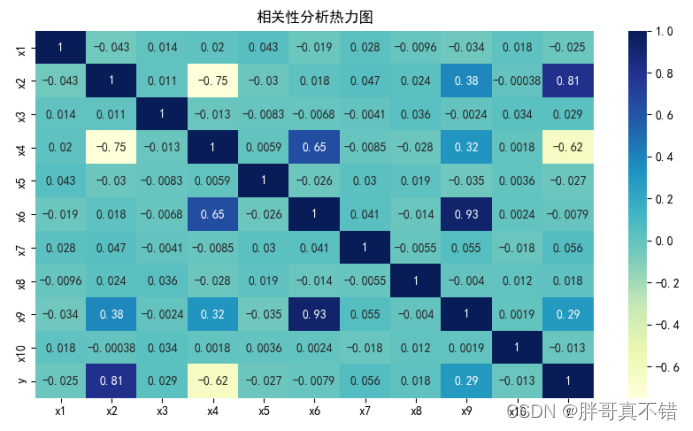
从上图中可以看到,数值越大相关性越强,正值是正相关、负值是负相关。
5.特征工程
5.1 建立特征数据和标签数据
关键代码如下:

5.2 数据集拆分
通过train_test_split()方法按照80%训练集、20%测试集进行划分,关键代码如下:
![]()
5.3 数据样本增维
数据样本增加维度后的数据形状:

6.构建SMA黏菌优化算法优化LSTM分类模型
主要使用SMA黏菌优化算法优化LSTM算法,用于目标分类。
6.1 SMA黏菌优化算法寻找最优参数值
最优参数:

6.2 最优参数值构建模型
|
编号 |
模型名称 |
参数 |
|
1 |
LSTM分类模型 |
units=best_units |
|
2 |
epochs=best_epochs |
6.3 最优参数模型摘要信息

6.4 最优参数模型网络结构
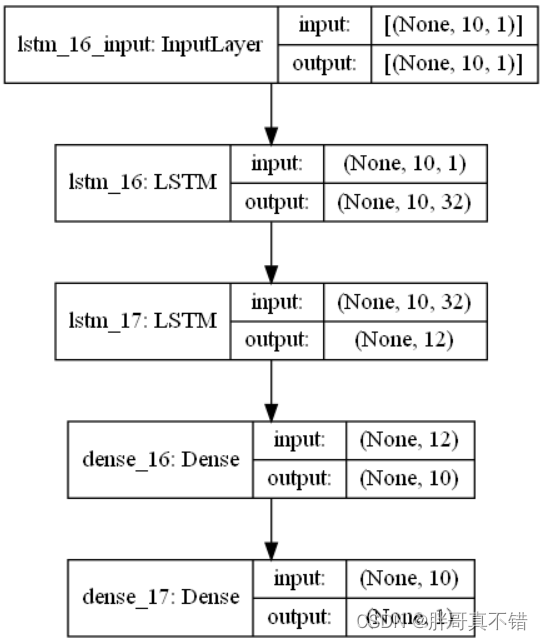
6.5 最优参数模型训练集测试集损失和准确率曲线图
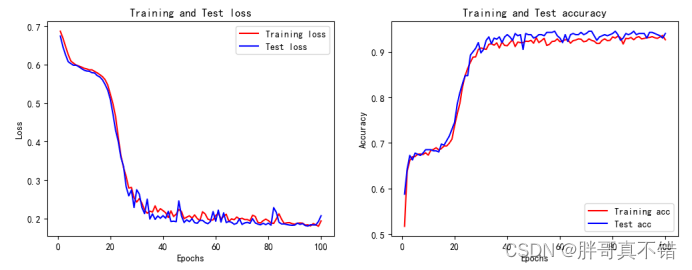
7.模型评估
7.1评估指标及结果
评估指标主要包括准确率、查准率、查全率、F1分值等等。
|
模型名称 |
指标名称 |
指标值 |
|
测试集 |
||
|
LSTM分类模型 |
准确率 |
0.9400 |
|
查准率 |
0.945 |
|
|
查全率 |
0.9356 |
|
|
F1分值 |
0.9403 |
|
从上表可以看出,F1分值为0.9403,说明模型效果良好。
关键代码如下:

7.2 分类报告
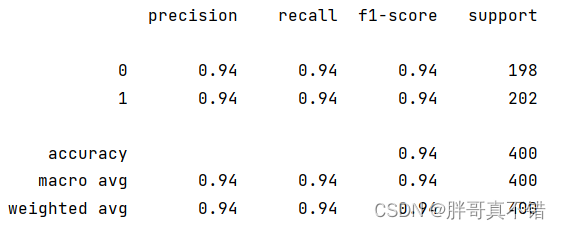
从上图可以看出,分类为0的F1分值为0.94;分类为1的F1分值为0.94。
7.3 混淆矩阵
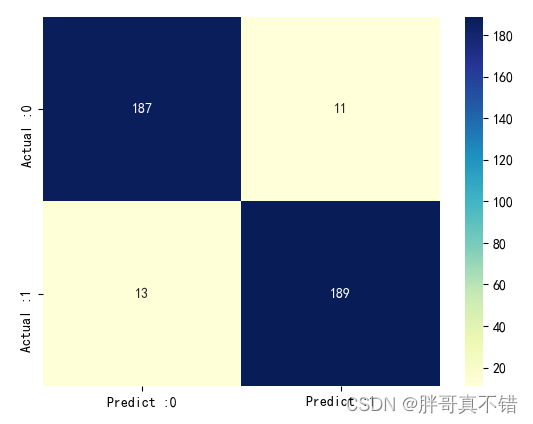
从上图可以看出,实际为0预测不为0的 有11个样本;实际为1预测不为1的 有13个样本,整体预测准确率良好。
8.结论与展望
综上所述,本文采用了SMA黏菌优化算法寻找LSTM分类算法的最优参数值来构建分类模型,最终证明了我们提出的模型效果良好。此模型可用于日常产品的预测。
更多推荐
 已为社区贡献58条内容
已为社区贡献58条内容







所有评论(0)