医学影像中用 python 读取 nrrd 文件、nrrd转nii、nrrd转h5
用 python 读取 nrrd 文件一般使用 pynrrd 这个库1 安装pynrrdpip 安装pip install pynrrd源码安装pip install git+https://github.com/mhe/pynrrd.gitcd pynrrdpip install .// 注意不要忘了最后的小点2 读取 nrrd 文件//先导入 nrrd 库import nrrddata_pat
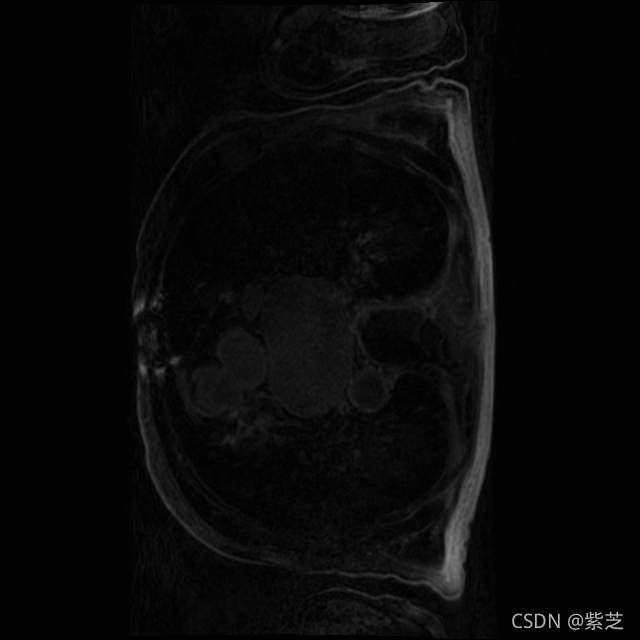

用 python 读取 nrrd 文件一般使用 pynrrd 这个库
1 安装pynrrd
pip 安装
pip install pynrrd
源码安装
pip install git+https://github.com/mhe/pynrrd.git
cd pynrrd
pip install . // 注意不要忘了最后的小点
2 读取 nrrd 文件
//先导入 nrrd 库
import nrrd
data_path=r'你自己的 nrrd 文件路径’
data,options=nrrd.read(data_path) # 读入 nrrd 文件
'''
data:保存图片的多维矩阵;
nrrd_options:保存图片的相关信息
'''
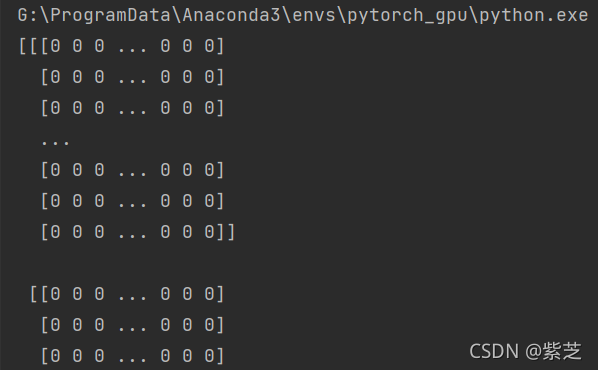
可以打印查看data 和 nrrd_options 里面的内容:
print(data)
print(options)
OrderedDict([('type', 'unsigned char'), ('dimension', 3), ('space', 'left-posterior-superior'), ('sizes', array([640, 640, 88])), ('space directions', array([[1., 0., 0.],
[0., 1., 0.],
[0., 0., 1.]])), ('kinds', ['domain', 'domain', 'domain']), ('encoding', 'raw'), ('space origin', array([0., 0., 0.]))])
3 查看 nrrd 文件的形状
# 查看nrrd文件的形状
print(data.shape) # (640, 640, 88)
正常情况下读取出来的 nrrd 数据应该都是类似这种的格式:[w, h, n]第三维度保存的是图像的序号;
但是在一些情况先我们读出来的数据格式是:[c, w, h, n] ;这乍一看没啥为题,就是多了第一维度的通道信息。
4 将 nrrd 矩阵转换为图像数据
4.1 数据格式为[w, h, n]
import nrrd
import PIL
from PIL import Image
data_path = './data/laendo.nrrd'
data, options = nrrd.read(data_path) # 读入nrrd文件
'''
data:保存图片的多维矩阵
nrrd_options:保存图片的相关信息
'''
print(data)
print(options)
# 查看nrrd文件的形状
print(data.shape)
img = Image.fromarray(data[:, :, 50])
temp_path = './data/temp_img.png'
img.convert('RGB').save(temp_path)
4.2 数据格式为[c, w, h, n]
我的 nrrd 文件转成图像后是用于深度学习,
data.shape = (3, 512, 512, 237)
所以就先将其转换为PIL 格式(需要用到PIL库)
import PIL # 测试PIL库是否安装成功
from PIL import Image
img=Image.fromarray(data[:,:,20]) # 截取第20张图片
究其原因,是因为[c, w, h, n]中,c的原因;如果要想成功的将其转换为图像数据,可以使用下面这条命令
img=Image.fromarray(data[1,:,:,20]) # 截取第一个通道的第20张图片
但是我经过对比,发现三个通道上的信息不同,如果随意选择一个进行提取,那么就会丢失另外两个通道的信息。后面我问其他人,他们说第一个维度上的数值还有为 2 的,瞬间就给我整懵了。
后来经过查询以及对三个通道的图像对比,怀疑第一维度上面的信息可能表示 患者接受扫描的期相,1 表示平扫,只有一期;2 表示增强,有动脉期和静脉期;3 表示造影,有动脉期、静脉期和平衡期。
同时从扫描角度考虑,在这几个扫描期相中,患者的位置是相对固定的,也就是说我们可以随意选择其中一个期相都可以;但是反过来,我们可以将所有期相的数据都利用上。
如果只是想随意选择其中的一个期相,使用上面一个命令即可,如果想将所有的期相都用起来,那么需要增加一个循环。
for j in range(img.shape[0]):
for i in range(img.shape[3]):
img=Image.fromarray(data[j,:,:,i]) # 截取第j个通道的前i张图
//循环结束后,就可以将所有数据都提取出来并直接转换为图片数据
//最后贴一个相对比较完整的代码
import PIL
import nrrd
import os
data_path=./data/PA1.nrrd # nrrd 文件的保存路径
save_path=./data/save # 图片数据的保存文件夹
# 检查路径
assert os.path.exists(data_path), data_path+' : path error !'
if not os.paht.exists(save_path):
os.makedir(save_path)
data, options = nrrd.read(data_path)
for j in range(data.shape[0]):
for i in range(data.shape[3]):
img=Image.fromarray(data[j,:,:,i]) # 截取第j个通道的前i张图
temp_path=save_path + '/' + str(j) + '_' + str(i) +'.png'
img.convert('RGB').save(tmp_path) # 保存图像数据
//循环结束后,就可以将所有数据都提取出来并直接转换为图片数据
5 nrrd转nii
import nrrd
import nibabel as nib
import numpy as np
# nrrd 文件保存路径
data_path=r'./data/1.nrrd'
save_path='./data/1.nii'
data,options=nrrd.read(data_path) # 读取 nrrd 文件
img=nib.Nifti1Image(data,np.eye(4)) # 将 nrrd 文件转换为 .nii 文件
nib.save(img,save_path) # 保存 nii 文件
6 nrrd转h5
- Download heart MRI data MICCAI 2018 Atrial Segmentation Challenge.
- Pre-processing data like existing work UA-MT
import numpy as np
from glob import glob
from tqdm import tqdm
import h5py
import nrrd
output_size = [112, 112, 80]
def covert_h5():
listt = glob('../../LA_dataset/2018LA_Seg_Training Set/*/lgemri.nrrd')
for item in tqdm(listt):
image, img_header = nrrd.read(item)
label, gt_header = nrrd.read(item.replace('lgemri.nrrd', 'laendo.nrrd'))
label = (label == 255).astype(np.uint8)
w, h, d = label.shape
tempL = np.nonzero(label)
minx, maxx = np.min(tempL[0]), np.max(tempL[0])
miny, maxy = np.min(tempL[1]), np.max(tempL[1])
minz, maxz = np.min(tempL[2]), np.max(tempL[2])
px = max(output_size[0] - (maxx - minx), 0) // 2
py = max(output_size[1] - (maxy - miny), 0) // 2
pz = max(output_size[2] - (maxz - minz), 0) // 2
minx = max(minx - np.random.randint(10, 20) - px, 0)
maxx = min(maxx + np.random.randint(10, 20) + px, w)
miny = max(miny - np.random.randint(10, 20) - py, 0)
maxy = min(maxy + np.random.randint(10, 20) + py, h)
minz = max(minz - np.random.randint(5, 10) - pz, 0)
maxz = min(maxz + np.random.randint(5, 10) + pz, d)
image = (image - np.mean(image)) / np.std(image)
image = image.astype(np.float32)
image = image[minx:maxx, miny:maxy]
label = label[minx:maxx, miny:maxy]
print(label.shape)
f = h5py.File(item.replace('lgemri.nrrd', 'mri_norm2.h5'), 'w')
f.create_dataset('image', data=image, compression="gzip")
f.create_dataset('label', data=label, compression="gzip")
f.close()
if __name__ == '__main__':
covert_h5()
更多推荐
 已为社区贡献15条内容
已为社区贡献15条内容







所有评论(0)