metaGEM工具:直接从宏基因组重建基因组尺度的代谢模型
宏基因组测序和基因组拼接可以在一个物种或菌株水平的分辨率上探索个性化的、环境特异性的微生物群落。通过重建宏基因组组装基因组(MAGs),揭示了很大程度的微生物种间和种内遗传多样性。Zelezniak团队于2021年在Nucleic Acids Research发表了metaGEM工具(https://github.com/franciscozorrilla/metaGEM),可以直接从宏基...
宏基因组测序和基因组拼接可以在一个物种或菌株水平的分辨率上探索个性化的、环境特异性的微生物群落。通过重建宏基因组组装基因组(MAGs),揭示了很大程度的微生物种间和种内遗传多样性。Zelezniak团队于2021年在Nucleic Acids Research发表了metaGEM工具 (https://github.com/franciscozorrilla/metaGEM),可以直接从宏基因组进行多物种群落进行基因组尺度的代谢模型建模。该管道自动化了从MAGs拼接,到构建原核基因组尺度的代谢模型以及群落水平的通量平衡分析(FBA)模拟的所有步骤。
metaGEM是一个集成了多种已有生物信息学和代谢建模工具的Snakemake工作流程,用于预测微生物群落中细菌群落内的代谢相互作用。通过整个宏基因组测序数据集,可以重建宏基因组组装的基因组(MAGs),然后将其转化为基因组尺度的代谢模型(GEMs)进行模拟。其他输出包括丰度估计、分类归属、生长速率估计、泛基因组分析和真核基因组组装的鉴定。
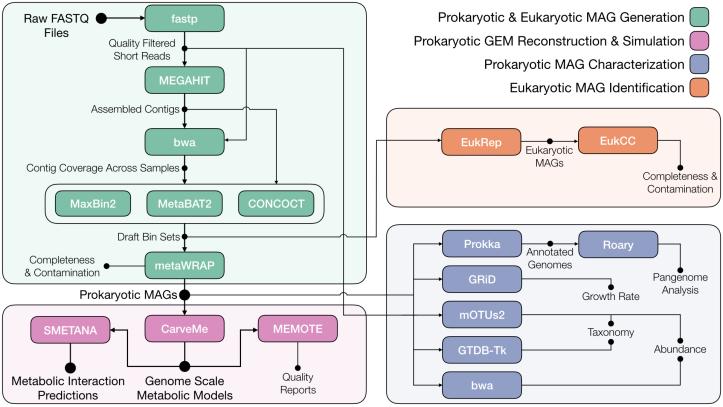
图1 metaGEM工具涵盖的工作流程
#1. metaGEM安装mamba create -n metagem -c bioconda metagem#从metaGEM的github主页克隆脚本并切换到工作路径git clone https://github.com/franciscozorrilla/metaGEM.git && cd metaGEM/workflow#2. metaGEM的运行Usage: bash metaGEM.sh [-t|--task TASK] #选择工作工具和任务[-j|--nJobs NUMBER OF JOBS] #任务数[-c|--cores NUMBER OF CORES] #线程数[-m|--mem GB RAM] #运行内存设置[-h|--hours MAX RUNTIME] #运行时间设置[-l|--local]-t参数设置选: fastp megahit, crossMapSeries, kallistoIndex, crossMapParallel, kallisto2concoct, concoct, metabat maxbin, binRefine, binReassemble, extractProteinBins, carveme, memote, organizeGEMs, smetana, extractDnaBins, gtdbtk, abundance#运行例子bash metaGEM.sh -t metabat -j 3 -c 60 -m 900 -h 12#核心工作流程:Quality filter reads withfastp #质控Assembly withmegahit #组装Draft bin sets withCONCOCT, MaxBin2, and MetaBAT2 #基因组拼接Refine & reassemble bins withmetaWRAP #基因组提纯、重组装Taxonomic assignment withGTDB-tk #基因组物种注释Relative abundances withbwa and samtools #基因组丰度定量Reconstruct & evaluate genome-scale metabolic models withCarveMe and memote #基因组代谢建模Species metabolic coupling analysis withSMETANA #物种代谢模型耦合分析Growth rate estimation withGRiD, SMEG or CoPTR #基因组生长速率预测Pangenome analysis withroary #泛基因组分析Eukaryotic draft bins withEukRep and EukCC #真核生物基因组草图拼接Reference:[1]Zorrilla F, Buric F, Patil K R, et al. metaGEM: reconstruction of genome scale metabolic models directly from metagenomes[J]. Nucleic Acids Research, 2021, 49(21): e126-e126.[2]https://github.com/franciscozorrilla/metaGEMReference:
[1]Zorrilla F, Buric F, Patil K R, et al. metaGEM: reconstruction of genome scale metabolic models directly from metagenomes[J]. Nucleic Acids Research, 2021, 49(21): e126-e126.
[2]https://github.com/franciscozorrilla/metaGEM
免责声明:本文仅用于学术分享,若有侵权,请联系邮箱:1372890765@qq.com删除或修改!
更多推荐
 已为社区贡献24条内容
已为社区贡献24条内容








所有评论(0)